目录
- 需要的库
- 读取dicom
需要的库
●Simpleitk
安装命令:conda install -c simpleitk simpleitk
使用:
import SimpleITK as sitk
●pydicom(不推荐,可能有些文件打不开)
安装命令:conda install -c conda-forge pydicom
●PIL
使用:from PIL import Image
读取dicom
读取Series信息
由于患者可以同时做多个检查,所以dicom中可以包含多个study(多次检查,一般只有一个检查,所以不用管这个)和series(多个部位,一些信息也是用不同series保存),所以第一步就是要读取series信息。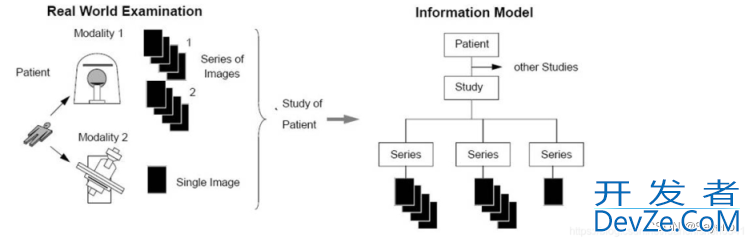
代码:
reader = sitk.ImageSeriesRead开发者_C开发er() ids = reader.GetGDcmseriesIDs(dict_name)
ids里包含了所有series的id,dict_name为dicom文件所在目录路径
读取Series中所有图片利用上面读取的series的id可以分开读取不同series。代码:f编程or i in ids:
series_file_name = reader.GetGDCMSeriesFileNames(dict_name, i)
series_file_name中包含某个series所有图片的路径,一般我们只需要图片数量最多的那个series。ids为series的id列表,dict_name为dicom文件所在目录路径。
(重点)读取meta data(元数据)
dicom文件包含大量的元数据,会指出与该次检查的相关信息,常用的有 Slice thickness(层厚), Pixel spacing(像素间距)可以根据需要读取相应的元数据。代码:file_reader = sitk.ImageFileReader()
file_reader.SetFileName(series_file_name[0])
file_reader.ReadImageInformation()
study_id = file_reader.GetMetaData("0008|1030")
series_file_name[0]
可以为任意dicom文件的路径,“0008|1030"为元数据的tags,详细的可以参考http://www.yuantk.com/toolkit_40.html ,或者直接搜索"dicom tag”
转换为图片并保存
代码:image = sitk.ReadImage(series_file_name[0])
image_data = sitk.GetArrayFromImage(image)[0]
image_data = normalization(image_data)
Image.fromarray(image_data).convert("RGB").save(image_save_path)
series_file_name[0]可以为任android意dicom文件的路径,使用simpleitk读取的数据已经进利用斜率和截距进行了线性变换无需二次处理。image_save_path为保存的路径,注意路径要包含文件名(例:‘./1.png’),这里的要重点关注normalization函数,该函数涉及窗口技术。
(重点)窗口技术
由于dicom存储的是CT值矩阵,由于各种组织结构或病变具有不同的CT值,因此欲显示某一组织结构细节python时,应选择适合观察该组织或病变的窗宽和窗位,以获得最佳显示,这里需要利用窗口技术进行转换。窗口技术涉及两个关键参数,窗位与窗宽。窗宽(WW):是CT图像上显示的CT值范围,在此CT值范围内的组织和病变均以不同的灰度显示,而CT值高于此范围的组织和病变,均以白影显示,不再有灰度差异;反之,低于此范围的组织结构,python均以黑影显示,也无灰度差别。窗位(WL):是窗的中心位置。公式:WW=CTmax-CTmin WL=(CTmax+CTmin)/2 CTmax = WW/2+WL CTmin = WL-WW/2
转换后的图片灰度值=(CT值-CTmin) / (CTmax - CTmin) * 255
代码:def normalization(data, window_level=-600., window_width=2000.): high = window_level + window_jswidth / 2 low = window_level - window_width / 2 data = (data - low) / window_width * 255 data[data > 255] = 255 data[data < 0] = 0 return data
window_level和window_width根据实际需要设定,data为simpleitk读取的数据
到此这篇关于python读取预处理DICOM文件方式的文章就介绍到这了,更多相关Python读取DICOM文件内容请搜索我们以前的文章或继续浏览下面的相关文章希望大家以后多多支持我们!





 加载中,请稍侯......
加载中,请稍侯......
精彩评论